PROCEDIMIENTO PARA MARCAR UN ACIDO RIBONUCLEICO, Y FRAGMENTOS DE ADN MARCADOS ASI OBTENIDOS.
Procedimiento para la fragmentación y marcaje uniforme con amplificación de señal de un ácido ribonucleico (ARN),
comprendiendo dicho procedimiento:
- fragmentar de manera no específica y químicamente el ARN con el fin de producir fragmentos que presentan un fosfato terminal en el extremo 3'',
- fijar un primer ligando a dicho fosfato terminal, y
- unir indirectamente un agente de marcaje a dicho primer ligando, en el que dicho primer ligando está unido a un primer antiligando, dicho primer antiligando está unido a un segundo ligando y el agente de marcaje es un segundo antiligando portador de por lo menos un marcador y puede reaccionar con dicho segundo antiligando
Tipo: Resumen de patente/invención. Número de Solicitud: W9902072IB.
Solicitante: BIO MERIEUX
AFFYMETRIX, INC.
Nacionalidad solicitante: Francia.
Dirección: CHEMIN DE L'ORME,69280 MARCY L'ETOILE.
Inventor/es: LAAYOUN, ALI, DO,DUC, MIYADA,CHARLES GARRET.
Fecha de Publicación: .
Fecha Concesión Europea: 14 de Octubre de 2009.
Clasificación Internacional de Patentes:
- C12Q1/68B2D
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Fragmento de la descripción:
Procedimiento para marcar un ácido ribonucleico, y fragmentos de ADN marcados así obtenidos.
Campo de la invención
La presente invención se refiere a un nuevo procedimiento para el marcaje del ácido ribonucleico (ARN) con amplificación de señal.
Antecedentes de la invención
El estado de la técnica muestra que existe una multiplicidad de procedimientos destinados al marcaje de los nucleótidos, oligonucleótidos o ácidos nucleicos; se hará referencia a los oligonucleótidos y ácidos nucleicos con el término polinucleótidos. Los polinucleótidos se pueden marcar durante la síntesis o mediante la incorporación de por lo menos un nucleótido marcado.
Un primer procedimiento comprende la adición de un marcador a la base, que puede ser una base natural o una base modificada. Un segundo procedimiento propone añadir el marcador al azúcar, ya sea esta última un azúcar natural o un azúcar modificado. Un tercer procedimiento se refiere a añadir un marcador al fosfato.
De hecho, un experto en la materia que deba marcar un nucleótido o un análogo de nucleótido o un ácido nucleico, tenderá a añadir el marcador a la base o al azúcar, lo que es más conveniente y ofrece más opciones. Esto es, además, lo que se concluye de estudiar gran número de documentos tales como EP-A-0.329.198; EP-A-0.302.176; EP-A-0.097.373; EP-A-0.063.879; US-A-5.449.767; US-A-5.328.824; WO-A-93/16094; DE-A-3.910.151 y EP-A-0.567.841 en el caso de la base o EP-A-0.286.898 en el caso del azúcar.
El procedimiento que consiste en añadir el marcador al fosfato es más complejo especialmente debido a que los ácidos nucleicos son solubles en agua y la reactividad del fosfato en este medio es inferior en comparación con ésta en disolventes orgánicos.
Aún así, algunos documentos han propuesto procedimientos para marcar el fosfato. Esto implica, por ejemplo, al documento EP-A-0.280.058, que describe el marcaje de un nucleótido mediante la unión del marcador al fosfato, estando éste unido al azúcar en las posiciones 3' y/o 5', cuando se trata de un desoxirribonucleótido y en las posiciones 2', 3', y/o 5' cuando el nucleótido es un ribonucleótido. Este documento describe asimismo un polinucleótido u oligonucleótido que comprende por lo menos un nucleótido marcado tal como se describe anteriormente; dicho nucleótido se incorpora en el polinucleótido u oligonucleótido durante la síntesis.
Sin embargo, la estrategia de marcaje que se propone en el documento EP-A-0 280 058 no permite que se marquen los ácidos nucleicos uniformemente. La incorporación de los nucleótidos marcados en polinucleótidos no se puede controlar; depende completamente de la composición de los nucleótidos sintetizados. Así, algunos polinucleótidos pueden comprender un gran número de nucleótidos marcados mientras que otros pueden comprender ninguno en absoluto. En consecuencia, la intensidad de la señal emitida por tales ácidos nucleicos no es uniforme y por consiguiente será difícil interpretar los resultados cuando se detectan ácidos nucleicos.
En este caso, el marcaje se incorpora biológicamente sin control de las posiciones de los nucleótidos marcados.
El documento US-A-5.317.098 se refiere a ácidos nucleicos marcados en sus extremos 5'. Esta unión utiliza imidazol y un brazo conector. No existe una fragmentación asociada al marcaje. Además, el fosfato se añade al ácido nucleico y por consiguiente se utiliza una quinasa como procedimiento para introducir el fosfato, lo que conduce a por lo menos una etapa biológica adicional. Este documento describe el marcaje de oligonucleótidos 15meros. Cuando se utilizan ácidos nucleicos grandes en lugar de oligonucleótidos, este procedimiento conduce a la presencia de un marcador solamente en los extremos 5' y la actividad específica del ácido nucleico marcado es baja.
Además, cuando se realiza el marcaje en ácidos nucleicos grandes sin una etapa de fragmentación, también referida como etapa de escisión, la cinética de hibridación de estos ácidos nucleicos a sus secuencias complementarias es lenta lo que conduce a un rendimiento de hibridación bajo. Esto, por consiguiente, conducirá a una pérdida de señal cuantitativa y cualitativa. El impedimento estérico es un factor clave en esta reacción.
El impedimento estérico puede ser consecuencia no solamente de la longitud del ácido nucleico sino también de la existencia de estructuras secundarias. La fragmentación ayuda a romper (o reducir) estas estructuras y de este modo a optimizar la hibridación. El impedimento estérico tiene un papel particularmente importante en el caso de la hibridación a superficies sólidas que comprenden gran densidad de sondas capturadas, por ejemplo las "matrices" de ADN desarrolladas por la compañía Affymetrix, Inc. ["Accessing Genetic Information with High-Density DNA arrays", M. Chee, et al., Science, 274:610-614 (1996). "Light-generated oligonucleotide arrays for rapid DNA sequence analysis", A. Caviani Pease et al., Proc. Natl. Acad. Sci. USA, 91: 5022-5026 (1994, US nº 5.744.305, US nº 5.445.934). En esta metodología las sondas capturadas generalmente son de un tamaño reducido, de aproximadamente veinte nucleótidos.
En la materia se describe una multiplicidad de procedimientos para la fragmentación de ácidos nucleicos.
En primer lugar, la fragmentación puede ser enzimática, es decir, los ácidos nucleicos se pueden fragmentar mediante nucleasas (ADNasas o ARNasas) (Methods in Enzymol., vol. 152, S. Berger y A. Kimmel, ed. Academia Press, 1987, Enzymatic techniques and recombinant DNA technology, "Guide to molecular cloning", p91-110, Molecular cloning, a Laboratory Manual, J. Sambrook, E.F. Fritsch y T. Maniatis, Cold Spring Harbor Laboratory Press, 2nd Edition, P5.30-5.95, 1989).
Dependiendo del enzima implicado, esta reacción genera fragmentos pequeños o monómeros con un grupo hidroxilo o monofosfato en los extremos 5' o 3'.
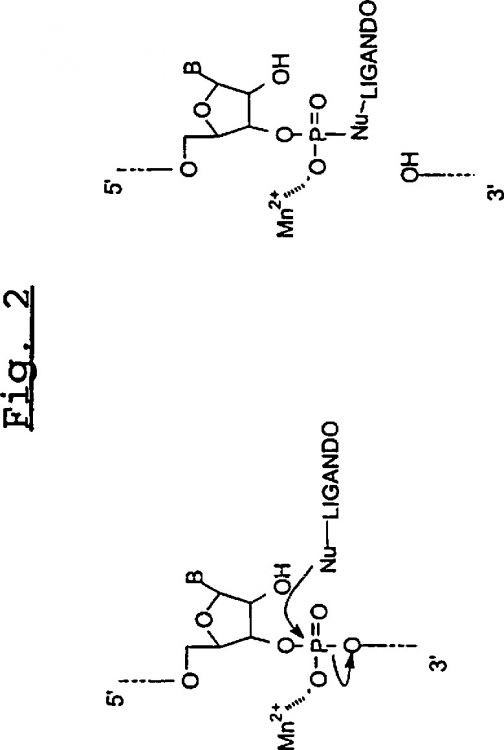
En segundo lugar, la fragmentación puede ser química. Por ejemplo, en el caso de secuencias de ADN, la depurinización o depirimidinilización utilizando agentes alquilantes genera lugares sin bases que a continuación se fragmentan en presencia de una base mediante un mecanismo referido como "eliminación-ß" (T. Lindahl et al., Rate of chain breakage at apurinic sites in double-stranded deoxyribonucleic acid., Biochemistry 11:3618-3623 (1972)). Los ADN se pueden fragmentar mediante mecanismos de oxidación, alquilación o adición de radicales libres, inter alia (M. Liuzzi et al., Characterization and damage in gamma-irradiated and OsO4-treated DNA using methoxiamine., Int. J. Radiat. Biol., 54:709-722 (1988)). Los cationes metálicos, que frecuentemente se combinan con moléculas orgánicas utilizadas como catalizadores, por ejemplo los imidazoles, se utilizan con el fin de fragmentar ARN. (R. Breslow y R. Xu, Recognition and catalysis in nucleic acid chemistry, Proc. Natl. Acad. Sci. USA 90:1201-1207 (1993); J. Hovinen et al., Imidazole Tethered oligonucleotides: Synthesis and RNA cleaving activity, J. Org. Chem., 60:2205-2209 (1995)). Esta fragmentación se realiza preferentemente en medio alcalino y genera fragmentos con extremos 3' fosfato.
Sin embargo, el objetivo de estas fragmentaciones no es el de facilitar o permitir el marcaje.
El documento WO-A-88/04300 propone un procedimiento de fragmentación y marcaje del ARN, que utiliza moléculas de ARN que poseen propiedades enzimáticas, es decir, ribozimas. La ruptura catalizada con estos ribozimas es secuencia específica y la reacción produce fragmentos de ARN con grupos hidroxilo (OH) en el extremo 5' y un monofosfato en el extremo 3'. El marcaje, que es un marcaje exclusivamente radioactivo, se realiza a continuación mediante la incorporación de un fosfato radiactivo añadido, derivado de una molécula de GTP. Es una actividad fosfotransferasa de esta categoría de ribozimas, es decir, una actividad quinasa. La unión de fosfato radiactivo tiene lugar solamente en el grupo hidroxilo del extremo 5' y no se utiliza ningún fosfato resultante de la fragmentación para unir el marcador a los fragmentos de ARN. Además, la fragmentación solamente se realiza mediante las ribozimas, lo que implica la existencia de una especificidad entre las ribozimas y los ácidos nucleicos diana que se cortarán. El fosfato actúa entonces como marcador.
La presente invención permite la unión del marcador al fosfato de un fragmento de ácido nucleico que se libera durante la...
Reivindicaciones:
1. Procedimiento para la fragmentación y marcaje uniforme con amplificación de señal de un ácido ribonucleico (ARN), comprendiendo dicho procedimiento:
- - fragmentar de manera no específica y químicamente el ARN con el fin de producir fragmentos que presentan un fosfato terminal en el extremo 3',
- - fijar un primer ligando a dicho fosfato terminal, y
- - unir indirectamente un agente de marcaje a dicho primer ligando, en el que dicho primer ligando está unido a un primer antiligando, dicho primer antiligando está unido a un segundo ligando y el agente de marcaje es un segundo antiligando portador de por lo menos un marcador y puede reaccionar con dicho segundo antiligando.
2. Procedimiento según la reivindicación 1, en el que las combinaciones de primer ligando/antiligando y segundo ligando/segundo antiligando se seleccionan de entre el grupo que consiste en biotina/estreptavidina, hapteno/anticuerpo, antígeno/anticuerpo, péptido/anticuerpo, azúcar/lectina y polinucleótido/polinucleótido complementario.
3. Procedimiento según la reivindicación 2, en el que los primer y segundo ligandos son el mismo.
4. Procedimiento según la reivindicación 2, en el que los primer y segundo ligandos son diferentes.
5. Procedimiento según la reivindicación 4, en el que el primer ligando es un derivado de la fluoresceína y el segundo ligando es un derivado de la biotina.
6. Procedimiento según la reivindicación 1, en el que el primer ligando es un derivado de la biotina y el agente de marcaje o el primer antiligando es un derivado de la estreptavidina.
7. Procedimiento según la reivindicación 1, en el que la fragmentación y la fijación se efectúan en una etapa.
8. Procedimiento según la reivindicación 1, en el que la fragmentación y la fijación se efectúan en dos etapas.
9. Procedimiento según cualquiera de las reivindicaciones 1 a 8, en el que la fijación del primer ligando al extremo 3' de un fragmento de ARN se efectúa haciendo reaccionar una función reactiva, que es portada por dicho primer ligando, al fosfato que se encuentra en la posición 2', en la posición 3' o en la posición 2'-3' del monofosfato cíclico, con respecto a la ribosa.
10. Procedimiento según cualquiera de las reivindicaciones 1 a 9, en el que la fragmentación de dicho ARN y/o la fijación del primer ligando al extremo 3' de un fragmento de ARN se efectúa haciendo reaccionar una función nucleófila, electrófila o haluro que es portada por dicho primer ligando al fosfato en la posición 2', en la posición 3' o en la posición 2'-3' del monofosfato cíclico, con respecto a la ribosa.
11. Procedimiento según la reivindicación 1, en el que la fragmentación química del ARN se realiza mediante cationes metálicos que pueden o no estar combinados con un catalizador químico.
12. Procedimiento según la reivindicación 11, en el que los cationes metálicos son iones Mg++, Mn++, Cu++, Co++ y/o Zn++ y en el que el catalizador químico consiste en imidazol, un análogo sustituido, por ejemplo N-metilimidazol, o cualquier molécula química que presenta una afinidad por el ARN y que sea portadora de un núcleo imidazol o un análogo sustituido.
13. Procedimiento según cualquiera de las reivindicaciones 1 a 12, en el que la fijación del primer ligando al extremo 3' de un fragmento de ARN se efectúa mediante la reacción de una molécula R-X con el fosfato que está unido a la posición 2', a la posición 3' o a la posición 2'-3' del monofosfato cíclico de la ribosa, en el que R consiste en el primer ligando y X es la función reactiva, seleccionada de entre el grupo que consiste en el hidroxilo, amina, hidracina, alcoxilamina, haluro de alquilo, haluro de fenilmetilo, yodoacetamida o maleimida.
14. Procedimiento según la reivindicación 13, en el que R-X se selecciona de entre el grupo que consiste en 5-(bromofluoresceína) y derivados de la yodoacetil biotina.
15. Fragmento de ARN marcado que es obtenible mediante el procedimiento según cualquiera de las reivindicaciones 1 a 14, que comprende en el extremo 3' un fosfato o un tiofosfato, un primer ligando, un primer antiligando, un segundo ligando y un agente de marcaje que es un segundo antiligando portador de por lo menos un marcador, en el que dicho primer ligando está unido a dicho primer antiligando, dicho primer antiligando está unido a dicho segundo ligando y dicho segundo ligando está unido a dicho segundo antiligando y en el que dicho primer ligando es la fluoresceína, dicho primer antiligando es un anticuerpo antifluoresceína, dicho segundo ligando es la biotina y dicho segundo antiligando es la estreptavidina marcada.
16. Fragmento de ARN según la reivindicación 15, en el que comprende de 10 a 150 nucleótidos.
17. Utilización de un fragmento de ARN según la reivindicación 15 ó 16 como una sonda para la detección de ARN y/o ADN o un fragmento de ARN y/o un fragmento de ADN.
18. Utilización de un fragmento de ARN según la reivindicación 15 ó 16 como una diana marcada que puede unirse a una sonda de captura.
Patentes similares o relacionadas:
 MÉTODO DE DETECCIÓN DE SECUENCIAS DE ÁCIDO NUCLEICO Y SUSTRATO PARA DETECCIÓN DE SECUENCIAS DE ÁCIDO NUCLEICO, del 6 de Abril de 2011, de HITACHI PLANT TECHNOLOGIES, LTD: Método de detección de secuencias de ácido nucleico para detectar la presencia de una secuencia de ácido nucleico diana particular que va a detectarse […]
MÉTODO DE DETECCIÓN DE SECUENCIAS DE ÁCIDO NUCLEICO Y SUSTRATO PARA DETECCIÓN DE SECUENCIAS DE ÁCIDO NUCLEICO, del 6 de Abril de 2011, de HITACHI PLANT TECHNOLOGIES, LTD: Método de detección de secuencias de ácido nucleico para detectar la presencia de una secuencia de ácido nucleico diana particular que va a detectarse […]
 METODOS Y COMPOSICIONES PARA LA DETECCION Y EL ANALISIS DE ACIDOS NUCLEICOS POR AMPLIFICACION DE SEÑAL, del 24 de Junio de 2010, de NATIONAL RESEARCH COUNCIL OF CANADA: Un sistema de detección de ácido nucleico que comprende:
una sonda oligonucleotídica aniónica marcada con un fluoróforo; y
un politiofeno catiónico crómico por afinidad […]
METODOS Y COMPOSICIONES PARA LA DETECCION Y EL ANALISIS DE ACIDOS NUCLEICOS POR AMPLIFICACION DE SEÑAL, del 24 de Junio de 2010, de NATIONAL RESEARCH COUNCIL OF CANADA: Un sistema de detección de ácido nucleico que comprende:
una sonda oligonucleotídica aniónica marcada con un fluoróforo; y
un politiofeno catiónico crómico por afinidad […]
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.